作者简介:李佳霖(1980-),女,山东烟台人,助理研究员,主要从事海洋微生物生态学研究.E-mail:jlli@yic.ac.cn
微微型蓝细菌是迄今发现最小也是最古老的光合自养生物,在海洋中分布极广且丰度较高,作为重要的初级生产者对全球碳循环和海洋食物网具有重要贡献。在长期进化中微微型蓝细菌形成简并基因组和高度多样性,其分子生态学的研究能够为理解生物的基因型、表型与生态型的关系以及生物的适应性进化提供关键科学依据。借助分子生物技术的发展,海洋微微型蓝细菌分子生态学近年来的研究揭示了其分布的时空变化特征、不同亚型微尺度分布差异及影响分布的主要环境因子,探讨了环境因子影响下基因组在功能和表达上传递的适应机制信息。通过进一步扩充和挖掘基因组信息,结合分布规律论证微微型蓝细菌在分子水平上的环境适应机制,是实现生态系统微微型蓝细菌功能模块精细化数值模拟的关键,也是未来研究的主要方向和重要内容。
Picocyanobacteria are the smallest and most ancient photosynthetic autotroph known to date with wide distribution and abundant density in the oceans. As the major contributor to primary production, marine picocyanobacteria have significant impacts on global carbon cycles and ocean food web. Given the picocyanobacterial genome streamlining and remarkable diversity during the long process of evolution, studies of their molecular ecology provide key scientific basis for relationships among genotypic, phenotypic and ecological variations, as well as for insights of biotic adaptation evolution in the context of environmental requirements. With the application of newly molecular biotechniques over recent years, our reports on the molecular ecology of marine picocyanobacteria have not only revealed their features of temporal-spatial distribution, fine distribution patterns of genetical subgroups and environmental factors controlling their variations, but also explored their adaptation mechanism derived from information about genomic functions and expressions. Accessing the further progresses in genomic information of these organism, coupled with the advances in the environmental adaptation mechanisms on molecular level given valuable information on distribution characteristics, it would be able to simulate the regulatory and metabolic network of picocyanobacteria in high resolution integrated into the framework of ecosystem model which is the main focus on the future perspectives in molecular ecology of picocyanobacteria.
微微型蓝细菌是指粒径介于0.2~2.0 µ m之间的蓝细菌(也称为蓝藻, Cyanobacteria), 属于微微型原核浮游植物(Prokaryotic Picophytoplankton)类群, 是迄今为止发现的最小的光合自养生物。微微型蓝细菌主要包括聚球藻和原绿球藻2类, 它们广泛分布于全球海洋的真光层中, 其中原绿球藻甚至可以生存在真光层以下通过吸收极微弱的蓝光进行光合作用[1, 2]。
海洋中微微型蓝细菌的丰度非常高, 在温带和热带海区可以达到106个/mL。全球海洋的年均丰度为(3.4~3.7)× 1027个[3], 夏季我国南海北部聚球藻和原绿球藻的丰度均值分别达到4.3 × 103个/mL和7.0 × 104个/mL[4]。通过对各个海区40 946个实测数据的模拟估算, 全球微微型蓝细菌的生物量达到2.3 × 1011 g· C, 其中聚球藻和原绿球藻分别占47%和53%[5]。作为光合自养生物, 微微型蓝细菌吸收CO2制造自身生长需要的有机物, 其巨大的生物量既说明微微型蓝细菌在全球碳循环中作为生物泵的关键固碳作用, 也体现了微微型蓝细菌作为初级生产者对整个海洋食物网和能量流的重要启动作用[6~8]。
蓝细菌是地球上最古老的光合生物, 出现于27亿年前, 经过漫长的生物进化过程, 形成适应不同海洋生境的各种生态型(Ecotype)。不同生态型在系统发育上是同一种, 即16S rRNA编码基因相似性> 97%, 其基因组岛(Genomic Islands)序列存在明显差别, 进而表现为不同生态型间功能蛋白和生理形态的差异。在氮限制的加勒比海和印度洋, 原绿球藻基因组岛中存在大量的硝酸盐和亚硝酸盐同化基因[9], 而在磷限制的马尾藻海摄取磷的蛋白编码基因数量明显增加[10]。微微型蓝细菌的基因组通常小于2.86 Mb, 编码蛋白约为1700~3200个, 远低于蓝细菌基因组平均值5.33 Mb, 而原绿球藻的基因组则更小。微微型蓝细菌成为研究生物基因型(Genotype)、表型(Phenotype)和生态型的模式生物, 其分子生态学的研究能够为生物进化和适应机制提供重要科学依据。
20世纪90年代以来, 流式细胞和分子生物技术使微微型蓝细菌的研究获得显著成果[11~13]。而近年来随着高通量培养和分选技术, 及全基因组和元基因组测序技术的发展, 微微型蓝细菌研究取得了新的发展。本文结合近年来的相关文献, 从分子生态学角度对微微型蓝细菌的系统发育、研究方法、分布特征和适应机制4个方面的研究现状进行综述, 并对未来的研究内容和方向提出建议。
通过16S rDNA序列分析, 微微型蓝细菌的主要类群聚球藻和原绿球藻位于系统进化树的相邻分支, 都起源于含藻胆蛋白的浮游植物物种。两者的区别是聚球藻以藻胆蛋白作为光捕获天线(Lighting-harvesting Antennae)进行光合作用, 而原绿球藻的藻胆蛋白只有感光作用, 其光捕获功能通过含类囊体膜蛋白的叶绿素蛋白复合物(Pcb)实现[14]。海洋中微微型蓝细菌及其在系统发育树上的位置如图1所示。
海洋聚球藻归类于聚球藻第5类(Cluster 5), 依据其核糖体DNA的G+C摩尔含量、光合色素特征及生长环境可以进一步分为3个亚类(5.1~5.3)。目前海洋中发现的聚球藻主要为5.1亚类, 分布于开放海域, GC含量为55%~62%, 以藻红蛋白为主要光捕获色素蛋白。该亚类的生物多样性极高, 包括广域分布的8个类型(I~VIII), 稀有类型(IX)和分布于特定海域的类型(如X~XVI、CRD1/CRD2、WPC1/WPC2等)[15]。5.2亚类分布于河口和近海的高盐区, GC含量为63%~69%, 含藻蓝蛋白, 其基因型存在明显的地理分布差异, 在不同海区会形成不同的进化分支[16]。5.3亚类的GC含量较低为47%~49%, 主要分布于半咸水和近海海域。
原绿球藻与聚球藻5.1亚类在进化树上形成同一分支, 两者更近缘。多数原绿球藻基因组在进化过程中舍弃非必要基因, 造成基因组减少和特化, 通过减小细胞体积、增加细胞比表面积及减少氮磷等营养需求在海洋贫营养盐环境中获得生存竞争优势[17]。根据生态分布特点、光适应特性及二乙烯基叶绿素b与a含量的比值(DV-chl b/DV-chl a), 原绿球藻分为高光(High Light, HL)适应型和低光(Low Light, LL)适应性2类, 分别分布于真光层的上层和下层。其中LLI型原绿球藻较为特殊, 虽然其DV-chl b/DV-chl a 值较高归类于LL型, 但其分布介于HL和LL型中间层, 在纵向混合较好的海域甚至可以与HL型共同分布于表层, 这说明LLI型对光强具有更广泛的适应性, 可能是一种新的光适应型(Phototype)原绿球藻[18]。 HNLC型也被定义为HLIII和HLIV型, 仅发现于高营养盐低生产力的铁限制型海域[19]。
流式细胞仪能够根据细胞大小和荧光特性对微微型蓝细菌进行定量和定性分析, 成为研究其在海洋中丰度分布的有效技术手段。近年来, 随着在灵敏性、可靠性和快速化方面的改进, 流式细胞仪能够实现船载实时监测, 克服了固定剂固定对样品测定结果准确度的影响[20]。利用流式细胞仪的分选系统, 可以将目标蓝细菌从样品中分离出来进行细胞结构及系统发育分析。但分选前进行的切向流过滤富集和固定保存过程会造成细胞的损失, 聚球藻的损失率可能高达65%, 从而造成测定结果与实际的偏差[21]。通过双激光通道对不同光合色素的甄别, 可以将聚球藻细分为富含藻蓝蛋白和富含藻红蛋白的2种类群进行分别研究, 进而说明不同色素组成所代表的进化方向上聚球藻的生态分布和种群结构特征[22]。此外, 在微微型蓝细菌研究中的主要方法还包括通过荧光显微镜进行计数, 通过高效液相色谱法研究其色素组成, 通过从环境样品中提取基因组构建DNA文库说明其种群结构特征。相对于上述较为成熟的方法, 目前微微型蓝细菌的分子生态学研究主要从培养和基因组方面发展新的技术。
培养技术对分子生物学研究具有重要意义, 一方面它可以提供准确的全基因组信息, 并以此作为分析元基因组结果的参考序列; 另一方面对生理特性和新陈代谢的研究是验证基因组编码蛋白功能的重要手段[23, 24]。近年来发展的高通量稀释培养法(High-throughput Dilution-to-extinction Culturing, HTC)根据海洋的寡营养环境, 通过将海水中的微生物群体稀释至痕量增加海洋微生物培养的可能性, 进而实现海洋微生物的批量培养。Biller等[26]针对光合生物对该方法进行改进, 将原位海水经1µ m滤膜过滤后18~20 ° C暗置21天, 再经0.1µ m滤膜过滤后配制成PRO3V培养基灭菌, 原绿球藻经稀释由流式细胞仪加入含1ml上述培养基的48孔板中, 每孔中细胞的终浓度为10个/ml, 20° C、光周期(光暗比L:D=14:10)及光强度20 µ mol Q/(m2· s)条件下培养20天。通过上述方法培养分离到3株原绿球藻, 并获得其全基因组信息。
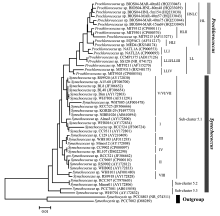 | 图1 海洋微微型蓝细菌的16S rDNA系统发育树(根据文献[12, 19, 25]序列信息构建)Fig.1 Phylogenetic tree of marine picocyanobacteria based on 16S rDNA (sequences derived from references[12, 19, 25]) |
单细胞基因组测序技术(Single-cell Genome Sequencing)提供了不依靠传统培养进行全基因组测序进而研究微生物多样性和系统发育的方法。该方法将环境中分离的单个细胞, 通过全基因组扩增(Whole Genome Amplification, WGA)获取足量的DNA模板, 并采用高通量测序获得全基因组序列信息[27, 28]。由于微微型蓝细菌的自发荧光特性利于分离获得单个细胞且基因组重复序列较少, 单细胞基因组测序技术在其分子生态学研究方面的应用前景广阔。Kashtan等[29]采用该方法研究了百慕大海域原绿球藻群落结构在不同时间的变化, 通过流式细胞仪的侧向角散射和自发荧光参数将原绿球藻的单个细胞分离至384孔板, 提取其DNA作为模板, 基于随机引物和φ 29 DNA聚合酶经多重置换扩增反应(Multiple Displacement Amplification, MDA)得到单细胞扩增基因组(Single Amplified Genome, SAG), 然后采用高通量测序技术获得单个细胞的基因组信息(全基因组覆盖率均值为70%)。研究结果表明原绿球藻在长期进化过程中形成数百个稳定的亚群, 各个亚群间基因组差异显著, 并由数个核心等位基因连接可变基因组成。
随着新一代测序技术的广泛应用, 微微型蓝细菌的全基因组和宏基因组数据得到快速积累和发布, 并形成相关数据库[30]。通过环境大样本的宏基因组测序和比较, 可以发现特定环境中微微型蓝细菌的代谢通路[31]。不同自然选择压力下微微型蓝细菌全基因组的比较则可以挖掘变异的受选择基因位点, 探索其生态适应机制[32]。借助于序列统计分析技术, 通过与生态位的环境参数相关联, 基因组数据不再仅仅描述高分辨率的系统发育组成和结构数据, 而且提供全面的基因型与表型和生态型的相关信息, 为研究未知的生物功能、代谢途径和生物进化提供科学依据[33]。
微微型蓝细菌的遗传多样性极高, 目前根据不同目标基因对微微型蓝细菌类群的研究结果, 至少将聚球藻主要亚类5.1划分为20类[15], 原绿球藻划分为12类[34]。16S rDNA序列是研究原核生物系统发育的指示基因, 可以用于鉴定聚球藻和原绿球藻的种属, 但由于其同源性> 97%, 难以提供足够的遗传信息用于微微型蓝细菌种内系统发育的研究。针对这一情况, 多个分子标记用于聚球藻和原绿球藻的分类研究(表1), 但目前还没有统一的指示基因。对海洋聚球藻的多位点进行分类学研究表明, 不同的目标基因可以从不同层面对聚球藻进行分类且结果存在差异, 而采用的同源性界值(Cut-off)标准也会对分类结果产生影响, 通过对不同分子标记结果进行相互印证能够实现聚球藻的分类细化[25]。
16S-23S rDNA内转录间隔区(Internal Trans- cribed Spacer, ITS)序列的变异性较大, 可用于研究遗传上接近的菌株间遗传进化关系, 是目前研究聚球藻和原绿球藻系统发育最常用的指示基因。对同一样品的原绿球藻群落, 采用ITS和单细胞全基因组进行遗传多样性分析, 结果表明两者构建的系统发育树拓扑结构相似, 说明ITS区可以准确标记16S rDNA序列高度相似的不同基因型原绿球藻[29]。
微微型蓝细菌的分布范围广泛, 覆盖全球海洋真光层直至真光层以下依然有极微弱蓝光的区域, 其生物量占整个海洋浮游植物的10%以上[5]。聚球藻的温度适应性强, 从热带到北极海域都有分布, 垂向最大深度一般为150m; 而原绿球藻多数分布于45° N至40° S之间的海域, 但其对光的捕获能力更强, 垂向可以分布至真光层下[35]。由于对营养盐的适应和竞争, 近海和上升流海域聚球藻数量相对较高, 而在远海的寡营养盐环境下原绿球藻构成优势种群[12]。时间尺度上, 聚球藻丰度在16° C季节达到峰值, 一般为105个/mL, 但在北冰洋常年低温环境下其季节变化不明显, 达到104个/mL[36]; 原绿球藻丰度最大值出现在26~28° C, 达到104个/mL, 由于对温度敏感, 其季节性变化较为明显, 一般在夏季达到极高值而冬季达到极低值[37], 海水温度低于10 ° C的环境中难以检测到原绿球藻。
| 表1 海洋微微型蓝细菌系统发育分析常用目标基因 Table 1 Target genes used in phylogenetic analysis of marine picocyanobacteria |
在长期自然选择压力下, 微微型蓝细菌形成了多个不同基因型类群, 而由于微微型蓝细菌个体小基因组简单, 对环境的响应调节及竞争能力有限, 因此各种类群有其特定的优势分布区域, 如图2所示[12]。聚球藻在海洋中主要亚类5.1的基因型类群多达20种, 目前基因型与表型和生态型的关系还不确定, 但研究结果已经证实I~IV型类群在海洋中有各自偏好的生态环境:I、IV型分布于南北纬30° 以上的高纬度近海海域, II型常见于热带和亚热带近海和远海海域, 而III型主要分布于寡营养环境的开放大洋[38]。V~VII型分布范围较广, 但是丰度一般较低, 只有在上升流海域形成优势类群。对聚球藻的现场调查和培养实验表明, 温度是造成其I/IV 和II型纬度分布差异的主要原因, I/IV型更适应于7~17° C水温的海域, 而II型在水温21~28° C海域丰度较高[39, 40]。原绿球藻类群的分布呈现垂向差异, HL型主要分布于上层, LL型主要分布于下层, 但在深层水混合作用强的海域LL型原绿球藻的丰度降低, HLII型形成优势种[41]。HLI和II型分布海域的差异由温度决定, HLI型最大生长温度为15° C决定其主要分布于温带海域(30~48° N, 35~40° S), HLII型最大生长温度为24~26° C主要分布于热带和亚热带海域(南北纬30° 间)。在吕宋海峡深海无光区也发现了HL II型原绿球藻的存在, 其分布深度可达500 m, 并在300 m处大量存在, 可能与其可以吸收有机物营异养生活有关[35]。原绿球藻可以吸收葡萄糖进行兼养生长模式的发现也证实了其在深水无光区分布的可能性[42]。
近年来, 微微型蓝细菌生态分布研究正从大尺度远洋考察扩展至对近海典型性海域的关注, 以期探索特殊生境下的新类群并对分布特征和规律进行更细致的描述。在大西洋西南部的阿根廷海(38~48° S), 16S rRNA的DGGE图谱表明, 秋季(3月)聚球藻以I/IV型类群为主, 且I类群分布范围更广, 这种分布类型与该海域水温为10~15° C的条件相符[48]。红海地处热带海域, 盐度极高而洋流条件特殊, 对该海域原绿球藻的研究表明, HLII类群在各深度(除100 m外)水层均为优势类群, 而在500 m的水层中亦发现HL II类群的存在, 这可能与红海深层水终年处于22° C高温有关, 说明HLII类群在代谢和光适应方面的特殊性仍有待于探索[41]。在亚德里亚海南部, 随着寡营养海水的流入微微型蓝细菌丰度增加, 聚球藻和原绿球藻在不同的低营养盐条件下丰度达到全年极大值, 并形成与微微型真核生物的竞争优势[49]。热带上升流海区的微微型蓝细菌多样性较高, 常发现新类群[43], 哥斯达黎加圆突区的聚球藻丰度达106个/mL, 对该区域聚球藻多样性研究发现其分布具有微尺度结构差异[47]。
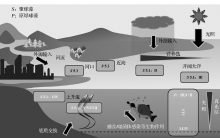 | 图2 海洋微微型蓝细菌的生态分布特征及其主要影响因子示意图(根据文献[13]修改)Fig.2 Conceptual diagram showing ecological patterns of marine picocyanobacteria, and representative environmental factors influencing their distribution (modified after reference [13]) |
我国近海海域微微型蓝细菌的生态类群和分布研究近年来也有相关文献报道。通过对ITS克隆建库对南海中尺度反气旋形成的上升流海域微微型蓝细菌的群落结构研究表明, 聚球藻类群(5.1亚类中的II、III、VII、X、XV和XVI及5.2亚类)和原绿球藻类群(HLI/II和LLIV)均有分布, 优势类群为聚球藻II和HLII类群, 而聚球藻III、XV和XVI类群在光强变化显著的涡流区数量较多[50]。东海海域微微型蓝细菌的分布通过ITS高通量测序结果进行解析, 发现共27种不同基因型类群且呈现明显的时空差异, 其中聚球藻在时间尺度上夏秋季优势种群为5.1II类群, 冬春季以5.1I/IV型类群为主, 与水温条件的影响相符[51], 主要类群与以psbA为目标基因克隆建库研究的结果一致[46]。在香港近海海域, 微微型蓝细菌根据细胞色素藻蓝和藻红蛋白荧光相对强度分为2类, 研究不同进化方向的种群结构特征[21]。目前研究手段仍以调查分析为主, 通过宏基因组和元基因组数据从分子基因水平揭示分布规律和机制的研究还极少涉及。
环境因子通过改变生长和增殖速度, 控制扩散区域, 进而影响微微型蓝细菌在海洋中的分布。由于微微型蓝细菌种类繁多, 不同类群甚至同一类群不同藻株对环境因子的响应存在差异, 而且海洋中多种环境因子的协同作用明显, 因此环境因子对微微型蓝细菌分布的影响作用非常复杂[12]。通过统计学方法, 如丰度和环境因子的相关分析, 多样性和环境因子的冗余分析(Redundancy Analysis, RDA)或典型对应分析(Canonical Correspondence Analysis, CCA), 可以建立其与环境因子的对应关系, 进而阐明环境因子的影响作用(图2)。
在全球尺度上, 根据观测结果影响微微型蓝细菌丰度分布的主要环境因子是海水温度和有效光辐射(Photosynthetically Active Radiation, PAR), 两者对聚球藻和原绿球藻丰度可解释变异量分别为0.69和0.34, 除了10~20° C范围内聚球藻与温度呈负相关外, 总体上聚球藻和原绿球藻与温度、有效光辐射呈正相关关系[3]。对于区域性海洋, 除上述影响因子外, 营养盐的限值作用对微微型蓝细菌的丰度影响较大, 在寡营养盐海域进行的氮、磷加富实验论证了随着氮、磷营养盐加入聚球藻和原绿球藻丰度的变化[52, 53]。在一些特殊海域, 还出现铁限值的情况, 在铁加富试验中, 原绿球藻的代谢速率会明显增加。随着人为污染的加剧, 污染物对微微型浮游植物的影响也日益显著, 最近在多环芳烃对原绿球藻影响作用的实验中发现, 原绿球藻的DNA合成速率和细胞分裂速率会降低, 但该影响在海洋环境中的效果还需要对近海微微型蓝细菌的分布进行进一步的研究进来论证[54]。此外, 洋流等物理条件造成的海水滞留时间变化、海水浊度、以及生物相互作用如捕食、噬菌体感染等, 都能够影响到微微型蓝细菌在海水中的丰度分布[6, 13, 55]。环境对微微型蓝细菌种群结构的影响则更为复杂, 与分类采用的目标基因和同源性界值直接相关, 以ITS分类的主要类群中温度和光照强度是主要的影响因子, 随着更多功能基因的应用和分类的细化, 与该基因相关的环境因子会对微微型蓝细菌类群分布产生影响[25]。
微微型蓝细菌对环境的偏好是长期进化的结果, 通过分子生物学的研究, 不仅能够了解其在全球尺度的分布特征和规律, 而且借助比较基因组、比较转录组学等后基因组学(Post-genome)的方法能够从基因水平上揭示其对环境适应的机制[56]。
光是浮游植物能量的来源, 昼夜节律的存在决定了浮游植物对光吸收具有适应机制, 微微型蓝细菌对光适应的研究以生理结构为基础, 随着近年来基因组学分析方法和基因组数据库的完善, 揭示了对光的适应机制及其差异。相较于聚球藻的藻胆蛋白体光吸收系统, 原绿球藻的Pcb整合于细胞膜上有利于能量的吸收和传递, 而且较大的比表面积可以增加蓝光的吸收量, 原绿球藻吸收光转化为能量的速率明显高于聚球藻, 因此在海水中原绿球藻的垂向分布范围较大, 而聚球藻的垂向分布极值一般为150 m[1]。聚球藻和原绿球藻在对日光照射的反映及适应方面也表现出明显的差别:①光损伤修复, 聚球藻的活性氧解毒酶基因、DNA修复基因如crtR和ocp表达量达到最大值, 而原绿球藻的基因线性化过程中丢失了大量的光损伤修复基因, 其应对机制可能通过改变代谢过程或启动保护机制来实现; ②光合反应, 在光强增加的过程中, 聚球藻光合作用的相关基因如光合系统I核心基因psaB、光捕获基因、glgA编码基因等的表达量升高, 而原绿球藻的相关基因则光强增加的最初数小时表达量升高, 随后呈现下降趋势; ③光响应机制, 聚球藻在日周期节律基因(如kaiA等)作用下表现出随光照强度变化的生物钟调节, 原绿球藻则表现出光压力下的负反馈调节[18, 57, 58]。原绿球藻的不同类群对光照适应性表现出明显差异, 比较全基因组信息发现除原绿球藻核心基因外, HL型含共有基因(Shared Genes)数250个, 其中95个在LL型中未发现, LL型含共有基因90个, 其中50个在HL型中未发现[59]。聚球藻类群较多, 在光适应方面的研究较少, 最近发现不同类群对温度表现出不同的生理特性, 相对应的基因组学差异需进一步研究[39]。
对营养盐的竞争是微微型蓝细菌在海洋寡营养盐环境下形成的适应性进化。在开阔海域的极端寡营养盐环境下, 细胞较小的浮游植物有竞争优势[17], 原绿球藻通过丢弃重复和垃圾基因, 甚至一些关键的但非必要基因如氧气生成过程中的重要基因氧化应激基因(Oxidative-stress Genes), 以减小基因组、增加比表面积来提高营养盐摄取速率[60]。针对特定海域的营养盐限制, 微微型蓝细菌通过获取相应的功能性基因提高对营养盐的吸收和转化。通过元基因组数据的比较, 发现在磷限制的北大西洋百慕大海域, 原绿球藻基因组中普遍含有磷吸收和转化的相关基因, 如phoA的基因覆盖率达到100%, 而在非磷限制海域仅为3%[10, 61]。在磷酸盐较低的海域, 以磷脂和次磷酸盐作为替代的新代谢途径的相关基因如phnYZ、phnD也在原绿球藻基因组中发现[31, 62]。原绿球藻的这类功能基因位于基因岛, 通过水平基因转移获得并依靠自然选择中的优势而稳定存在。对耐低磷环境的原绿球藻藻株MED4研究发现在磷限制胁迫下可以利用5′ -核苷酸酶吸收三磷酸腺苷(ATP)的磷酸集团, 而这一新代谢途径对应的基因信息还需要进一步研究解读[63]。微微型蓝细菌在对氮限制的适应上表现相似, 在氮限制海域其基因岛存在大量氮同化的相关基因nirB、narM、ntcA等[64], 表层海水的原绿球藻基因组中还发现了吸收尿素类降解产物氰酸酯的新代谢途径及其相关编码基因cynA[65]。
对聚球藻近海藻株CC9311的研究发现, 其对铜有较高的耐受力, 这与位于基因岛的sync_1495和sync_1217基因相关, 说明微微型蓝细菌能够针对所处的特定环境条件可能通过水平基因转移获得新的基因而产生适应机制[66]。CC9311在丝裂霉素C胁迫下recA、lexA和umuC等应激反应基因表达量升高, 也证实了微微型蓝细菌存在对环境变化的分子适应性机制[67]。在全球变化和人类活动影响下, 微微型蓝细菌在环境压力下表现出的这种基因和表达上的适应性变化, 对其生物多样性和自然选择的影响还需要进一步的研究探讨。
近年来微微型蓝细菌分子生态学借助于分析和测定技术的发展, 在系统发育、生态分布和适应机制方面的研究取得阶段性成果, 从分子水平解答了其从生理到进化方面的诸多科学问题, 未来研究的主体内容集中在以下3个方面:
(1)微微型蓝细菌基因组数据库的扩充、积累和挖掘:全基因组和元基因组信息是分子生物学研究的基础, 通过稀释培养法、单细胞基因组测序法能够提供微微型蓝细菌准确的全基因组信息, 以此为参考序列对环境样品的宏基因组信息进行解读, 可以剔除嵌合及冗余序列的干扰, 进而准确解析基因和基因组在遗传、调控和表达上传递的信息。
(2)微微型蓝细菌对环境条件的适应机制:适应机制一方面体现在不同类群对生态位的选择上, 包括典型和特殊海洋环境中微微型蓝细菌的种群结构特征及相应环境因子产生的影响; 另一方面体现在环境压力的变化对微微型蓝细菌基因型和表型产生的影响, 包括功能蛋白的表达和调控, 以及由此形成的生态功能的变化。通过对上述内容的研究, 能够对微微型蓝细菌的生态分布形成规律性认知, 从分子水平上探讨其对环境变化的适应策略。
(3)微微型蓝细菌生态功能模块的精细化模拟和嵌入:将微微型蓝细菌的规律性认识应用于数值模拟, 实现对其分布、变化和功能的精细化模拟和预测, 有助于探索其对全球海洋中的生态贡献。以此为基础功能模块在生态系统模型中实现嵌入, 可以更有效地研究海洋在全球物质和能量循环中的作用, 也有助于预测全球变化及人类活动对种群的影响为海洋环境开发和保护提供依据。
在基因、基因组等生物信息学基础上, 结合宏观上的环境参数和同位素标记的生态功能参数、微观上的细胞生理生化和蛋白结构指标, 开展微微型蓝细菌的分子生态学研究, 有助于认识生物多样性的本质, 理解生物与环境及生态系统的相互作用和反馈机制, 为更好的探索和利用海洋资源提供必要的科学支持。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
|
| [54] |
|
| [55] |
|
| [56] |
|
| [57] |
|
| [58] |
|
| [59] |
|
| [60] |
|
| [61] |
|
| [62] |
|
| [63] |
|
| [64] |
|
| [65] |
|
| [66] |
|
| [67] |
|


